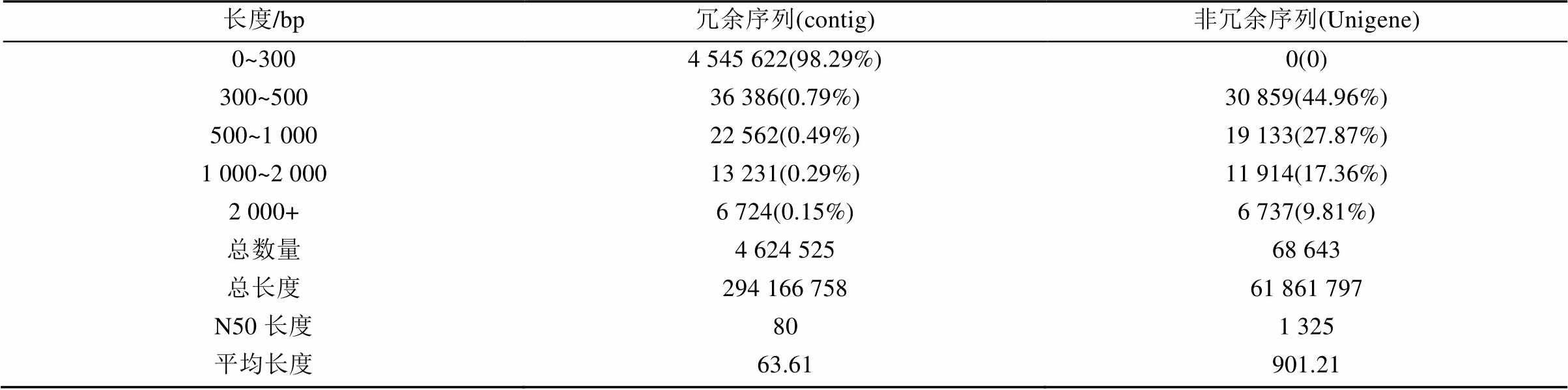
表1 青叶胆转录组组装结果统计
Tab. 1 Summary of the transcript generated fromS. mileensis
吴昕怡,严媛,刘小莉*
(云南中医学院中药学院,昆明 650500)
摘要:目的探讨青叶胆中獐牙菜苦苷生物合成的遗传基础。方法采用Illumina Hiseq 2500高通量测序技术,对青叶胆全草进行转录组测序。结果共获得68 643个unigene,平均长度901 bp,共有42 954条unigene注释到NR,SwissProt,COG,KOG,GO,Pfam数据库中,獐牙菜苦苷生物合成的3个阶段中,有43条unigene参与中间体的生成,3条unigene参与萜类骨架合成,25条unigene可能参与萜类最后的修饰。结论首次对青叶胆转录组进行测序分析,获得了可能参与獐牙菜苦苷生物合成的候选基因,为后续功能基因的挖掘奠定了基础。
关键词:青叶胆;转录组;Illumina Hiseq 2500;獐牙菜苦苷
青叶胆(Swertia mileensisT. N. Ho et W. L. Shih),为龙胆科獐牙菜属植物,别名青鱼胆、苦胆草、肝炎草[1]。是中国药典2015年版中中药材青叶胆的唯一基源植物,具有清肝利胆、清热利湿的功效[2],是云南特有中药和民族药,仅分布于云南红河州的弥勒、开远等县,在哈尼族、彝族地区用于治疗肝炎等疾病具有悠久的历史[3]。随着青叶胆药用价值的发现,经过近20年的滥采滥挖,野生居群已极为少见,加上生境不断丧失和遭到破坏(如道路建设、开荒、开发沙场等),青叶胆已呈零散形分布,片断化明显[4]。近年来,对青叶胆的研究主要集中在化学成分与含量测定[5]、药理作用[6]、遗传多样性[7]、组织培养[8]以及可持续利用[9]等方面,对青叶胆的代谢途径和相关基因的研究,仍处于空白状态。青叶胆治疗肝炎的主要活性成分是獐牙菜苦苷,獐牙菜苦苷具有抗乙型肝炎病毒活性[10]、保肝、抗炎、镇痛、解痉和抑制中枢神经等作用[6]。
植物的代谢产物通常具有长而复杂的生物合成途径,并且有许多催化代谢转化的酶[11]。代谢途径的酶通常由大基因家族编码,通常不能基于序列分析严格预测单个基因的特定功能[12]。转录组数据挖掘是发现编码涉及各种代谢途径的酶的基因或基因家族的有效方式。随着高通量测序技术的不断发展,测序成本降低,获得了丰富的数据,为转录组数据研究和分析提供了便利。近年来,国内外已经开展了人参[13]、铁皮石斛[14]、丹参[15]等多种药用植物的转录组研究,转录组研究已经成为挖掘功能基因的重要手段之一。基于此,本研究采用Illumina Hiseq 2500高通量测序技术,对青叶胆转录组进行测序分析,并应用生物信息学方法对所得EST序列进行拼接与组装,结合生物信息学的方法对转录组数据库序列进行功能注释、功能分类和代谢通路分析,搜索获得獐牙菜苦苷生物合成途径中的候选基因,同时研究了候选基因的表达,为青叶胆基因组水平上的研究奠定基础,同时也为后续青叶胆功能基因的研究提供数据。
采自云南省红河州弥勒市的野生青叶胆全草,酒精棉擦拭干净,经液氮速冻后,保存于-80 ℃中待用,设置3个生物学重复。
按照Takara Minibest Universal RNA Extraction Kit试剂盒提取青叶胆全株RNA,分别采用Nanodrop、Qubit 2.0、Aglient 2100方法检测RNA样品的纯度、浓度和完整性等,保证使用合格的样品进行转录组测序。质检合格后,委托百迈克生物科技有限公司完成转录组测序,测序平台为Illumina Hiseq 2500,测序读长为PE125。
测序得到的原始数据中会有极少部分的Reads带有测序引物、接头等人工序列,需要将其从Reads中截除,同时需要过滤低质量值数据,确保数据质量,经过上述一系列的质量控制之后得到的高质量Reads,称之为Clean Data。利用Trinity软件对Clean Data进行组装,首先将测序Reads打断为较短的片段(K-mer),然后将这些小片段延伸成较长的片段(Contig),并利用这些片段之间的重叠,得到片段集合(Component),最后利用De Bruijn图的方法和测序Read信息,在各个片段集合中分别识别转录本序列。
使用BLAST软件将获得的青叶胆Unigene序列与非冗余蛋白数据库(Non-repdundant protein database,Nr)、Pfam数据库(Protein family)、基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)、蛋白直系同源簇数据库(Clusters of Orthologous Groups,COG)、基因本体论数据库(Gene Ontology,GO)等数据库进行比对,使用KOBAS 2.0得到Unigene在KEGG中的KEGG Orthology结果,预测完Unigene的氨基酸序列之后使用HMMER软件与Pfam数据库比对,获得Unigene的注释信息。
经过测序质量控制,共得到15.09 Gb Clean Data,Q30碱基百分比≥96.64%。组装共得到242 701 315条Transcript和68 643条Unigene,Transcript与Unigene的N50分别为1 477.73 bp和1 325 bp,组装完整性较高。通过Mapped Reads在各Unigene上的位置分布,模拟mRNA片段化结果,检验mRNA片段化的随机程度。结果表明mRNA片段化随机性较高,说明各样品不存在严重降解现象。
利用Trinity软件进行组装,获得4 624 525个contigs片段,序列信息达到了294 166 758 bp;其中长度0~300 bp的最多,有4 545 622条,比例达到了98.29%。分布特征符合Illumina测序的预期结果,能够为后续的数据组装提供原始数据。在此基础上进一步拼接后获得68 643个Unigen,序列信息达到了61 861 797 bp;其中长度300~500 bp的最多,有30 859条,占总数的44.96%;其次为长度在500~1 000 bp的,有19 133条,占总数的27.87%;长度在1 000~2 000 bp的,有11 914条,占总数的17.36%;长度在2 000 bp以上的最少,有6 737条,占总数9.81%;具体结果见表1。
表1 青叶胆转录组组装结果统计
Tab. 1 Summary of the transcript generated fromS. mileensis

注 :括号内为该长度所占比例。
Note : The proportion of the length was in parentheses.
通过选择BLAST参数E-value≤1e-5和HMMER参数E-value≤1e-10,最终获得42 954个有注释信息的Unigene,比对的数据库包括Nr、KEGG、COG、GO等,COG,GO,KEGG,KOG,Pfam,Swiss-prot,Nr数据库的Unigene注释数目分别为15 199,21 645,16 342,27 532,31 591,23 740,39 197条。
2.2.1 青叶胆Unigene的序列相似性分析 NR数据库是NCBI中的非冗余蛋白质数据库,包含了Swiss-Prot、PIR(Protein Information Resource)、PRF(Protein Research Foundation)、PDB(Protein Data Bank)蛋白质数据库及从GenBank和RefSeq的CDS数据翻译过来的蛋白质数据信息。将青叶胆的Unigene与Nr数据库进行比对,共有39 197条Unigene注释到Nr数据库,占总Unigene的57.08%,结果见图1,青叶胆的Unigene与中粒咖啡(Coffea canephora)同源的序列最多,有10 040条,占总数的25.61%;其次是芝麻(Sesamum indicum)有1 911条,占总数的4.88%;第三是大豆(Glycine max)有1 882条,占总数的4.80%;最少的是阳芋(Solanum tuberosum)有616条,占总数的1.57%;而与其他物种同源的有19 051条,占总数的48.60%。
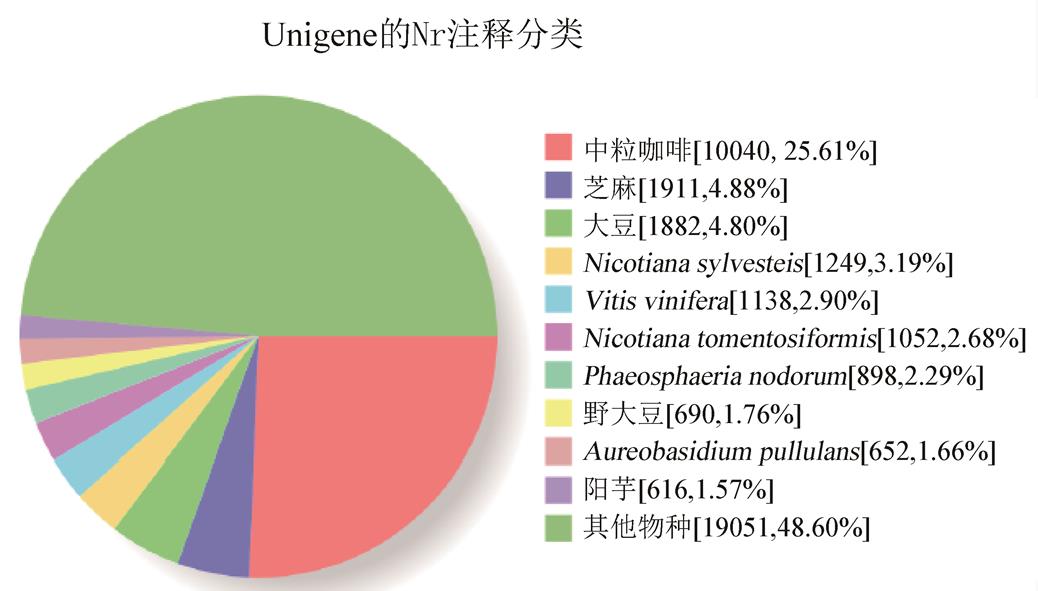
图1 Unigene的Nr注释分类
Fig. 1 Nr classification of assembled unigenes
2.2.2 青叶胆转录组Unigene的COG功能分类 COG(Clusters of Orthologous Groups)数据库是对基因产物进行同源分类的数据库,是一个较早的识别直系同源基因的数据库,通过对多种生物的蛋白质序列大量比较而来的。将获得的青叶胆Unigene与COG数据库进行比对,青叶胆Unigene按功能可分为25类(图2中用A~Z表示),Unigene涉及的COG功能类别较全面,一般功能预测类基因最多,有4 156条;其余依次为翻译、核糖体结构和生物合成类基因,有1 712条;翻译后修饰、蛋白翻转、分子伴侣类基因,有1 528条;复制、重组和修复,有1 510条;而核结构类基因最少,仅有3条;未发现胞外结构类基因。
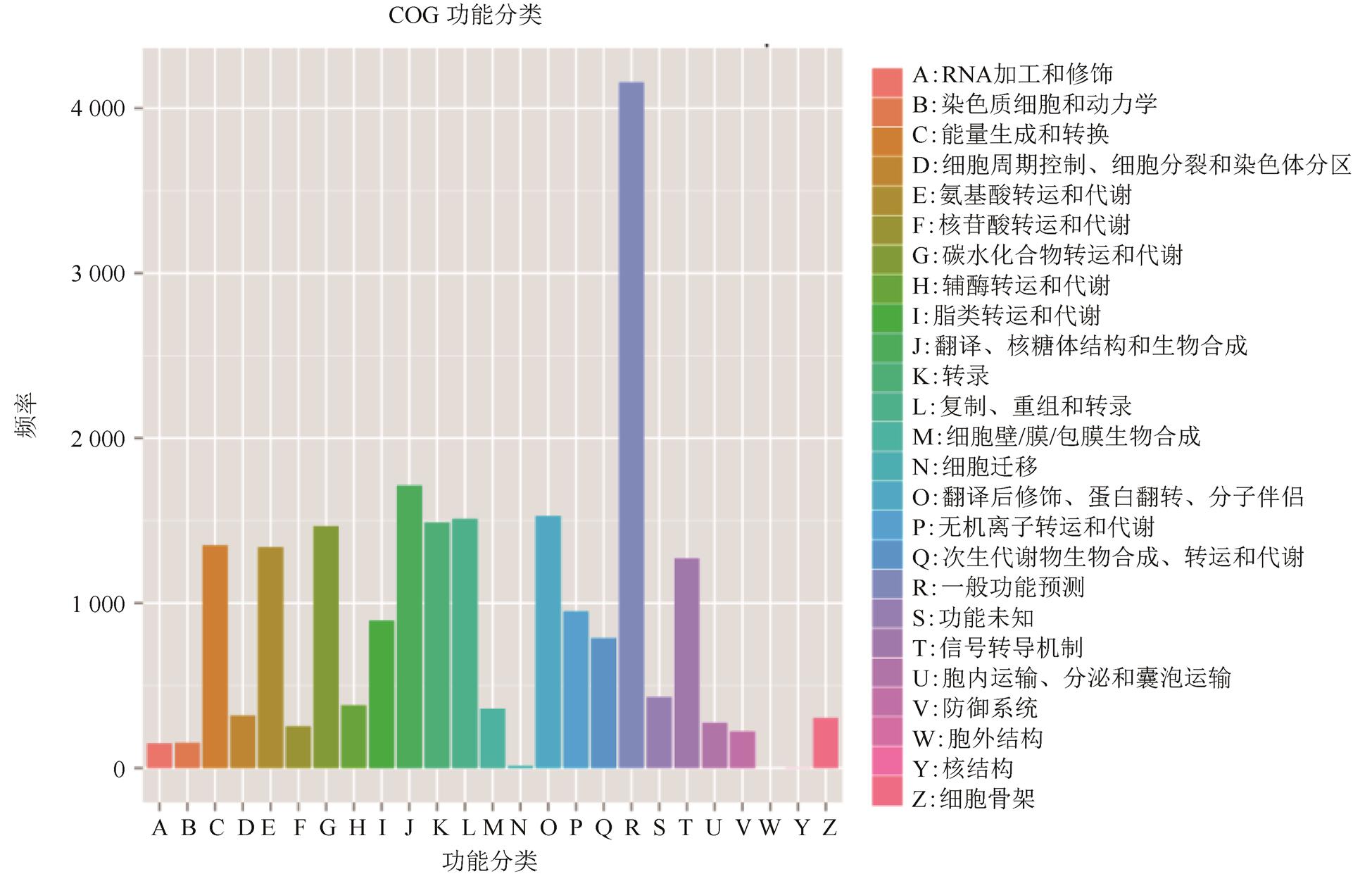
图2 Unigene的COG 功能分类
Fig. 2 COG classification of assembled unigenes
2.2.3 Unigene的GO功能分类分析 GO(Gene Ontology)数据库是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表来全面描述生物体中基因和基因产物的功能属性。该数据库总共有3大类,分别是分子功能(Molecular Function)、细胞组分(Cellular Component)和生物学过程(Biological Process),各自描述了基因产物可能行使的分子功能,以及所处的细胞环境和参与的生物学过程。将获得的Unigene与GO数据库进行比对,共有21 645条Unigene注释到GO数据库。青叶胆Unigene的GO功能分类分析具体结果如图3所示,从图中可以得知,青叶胆转录组的Unigene根据GO功能可分为3大类,包括细胞成分、分子功能、生物过程。生物过程这一大类涉及的基因最多,有55 239条;细胞成分这一大类涉及的基因次之,有39 139条;分子功能这一大类涉及的基因最少,有26 084条。进一步可将匹配序列富集为50个功能类别,其中最多的是代谢过程,有14 964条;其余依次是细胞过程,有12 232条;催化活性,有11 748条;而最少的是蛋白标签,仅有1条。
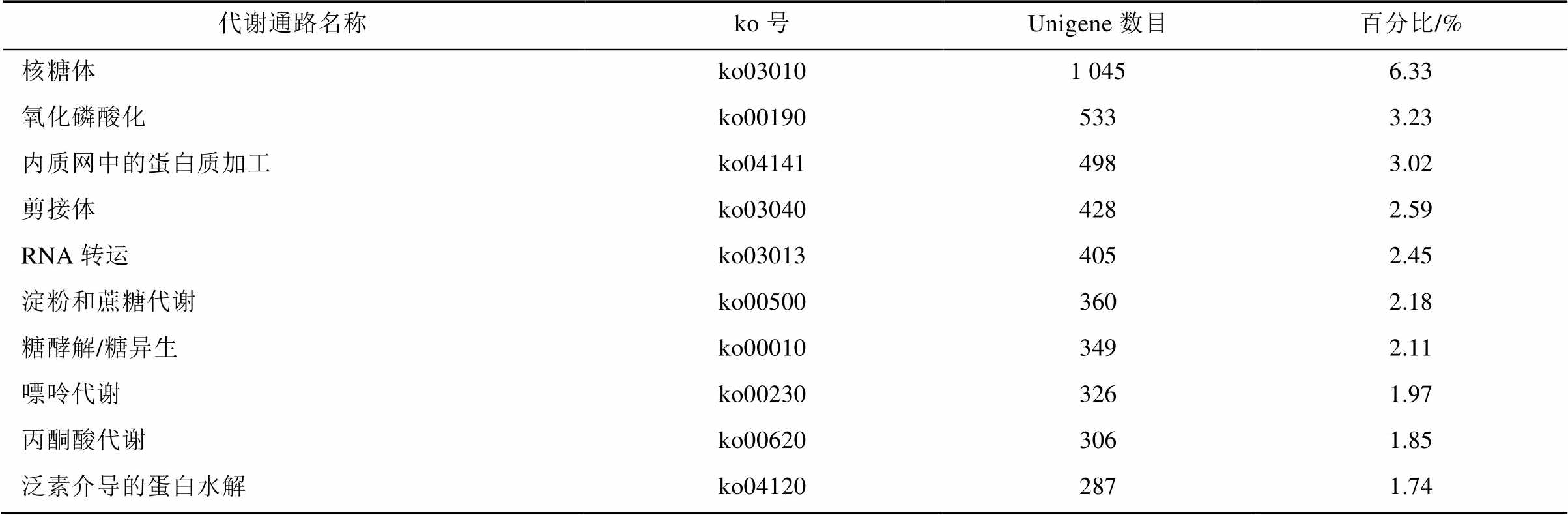
2.2.4 Unigene的KEGG功能分类分析 KEGG (Kyoto Encyclopedia of Genes and Genomes)数据库是系统分析基因产物在细胞中的代谢途径以及这些基因产物功能的数据库。它整合了基因组、化学分子和生化系统等方面的数据,包括代谢通路(PATHWAY)、药物(DRUG)、疾病(DISEASE)、基因序列(GENES)及基因组(GENOME)等。利用该数据库有助于把基因及表达信息作为一个整体的网络进行研究。将68 643个Unigene映射到KEGG数据库中,注释为代谢通路相关的序列共有16 516条,占总数的24.06%,共涉及到230种代谢途径,其中Unigene最多的是核糖体途径,共涉及到1 045条Unigene,占总数的6.33%;其次为氧化磷酸化途径,共涉及到533条Unigene,占总数的3.23%;第三为内质网中的蛋白质加工,共涉及到498条Unigene,占总数的3.02%。表2为Pathway富集性分析前10个通路的数据。
獐牙菜苦苷(swertiamarin)属于环烯醚萜类(secoiridoids)化合物,是由环烯醚萜类物质在C7、C8处开环演变而来,是特殊的单萜类化合物。目前青叶胆獐牙菜苦苷完整生物合成途径尚未完全明确,但地黄(Rehmannia glutinosa)[16]、川西獐牙菜(Swertia mussotii)[17]等植物中环烯醚萜类合成途径以及龙胆苦苷(gentiopicroside)[18]的生物合成途径具有较好的参考性。与萜类化合物的生物合成类似,环烯醚萜类化合物生物合成也可分为3个阶段,即中间体生成、萜类骨架合成和最后的修饰。为搜索参与獐牙菜苦苷生物合成相关的Unigene,将其映射到萜类生物合成途径中,发现有77条Unigene参与萜类骨架生物合成,其中有43条Unigene编码中间体生成的2条途径中的14个关键酶,1条为甲羟戊酸途径(MVA pathway),该途径在胞质中进行,有共计6种酶的参与;另一条途径为甲基-D-赤藓醇-4-磷酸途径(MEP pathway),它在质体中进行,有共计8种酶的作用,2个途径最终均合成IPP和其异构体DMAPP。同时也发现了催化IPP和DMAPP缩合成香叶基焦磷酸(GPP)的关键酶基因,即香叶基焦磷酸合酶(GPPS),具体结果见表3。
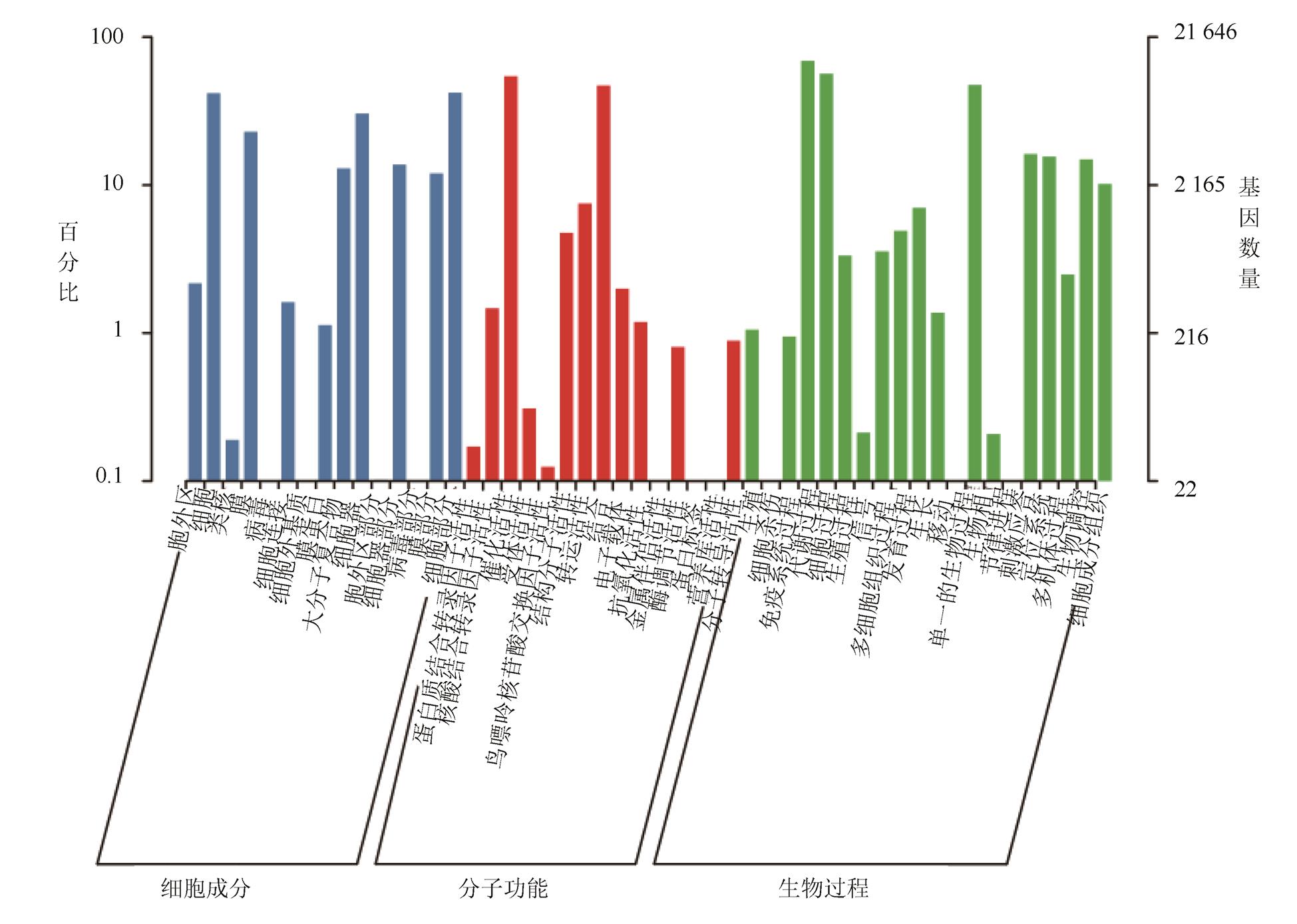
图3 Unigene的GO分类
Fig. 3 Gene ontology classification of assembled unigenes
表2 包含Unigene数量最多的10个代谢通路
Tab. 2 Top ten metabolic pathways involvingunigenes
GPP在香叶醇合酶(geraniol synthase,GES)作用下形成香叶醇,香叶醇C-2位置在香叶醇-10-羟化酶(Geraniol 10-hydroxylase,G10H)作用下生成10-脱氧香叶醇,10-脱氧香叶醇被氧化,在NAPDH氧化还原酶存在时,生成10-氧香叶醛,接着在环烯醚萜合酶(Iridiod synthase,IS)的催化下形成琉蚁二醛(iridodial),经过不断反应形成7-脱氧番木鳖酸(7-deoxyloganic acid),在7-脱氧番木鳖酸7-羟化酶(7-deoxyloganin 7-hydroxylase,DL7H)作用下生成马钱苷酸,在马钱酸甲基转移酶(loganic acid methyltranseferase,LAMT)的催化作用下生成番木鳖酸,裂环马钱子苷合酶(secologanin synthase,SLS)催化番木鳖酸形成裂环番木鳖酸(secologanin)。裂环番木鳖酸经若干步反应,最终形成獐牙菜苦苷。
表3 青叶胆环烯醚萜生物合成相关候选基因
Tab. 3 The candidate genes involved in ridoid biosynthesis identified inS. mileensis

通过查询Unigene在Swissprot与Nr数据库中的注释信息,结合KEGG代谢通路分析,发现了25条Unigene可能编码獐牙菜苦苷后修饰阶段的酶基因,有1条可能编码GES的Unigene,2条可能编码G10H的Unigene,4条可能编码10HGO的Unigene,1条可能编码IS的Unigene,3条可能编码DL7H的Unigene,3条可能编码LAMT的Unigene,11条可能编码SLS的Unigene。以上獐牙菜苦苷生物合成途径相关Unigene的获得,为后续深入研究青叶胆中獐牙菜苦苷生物合成途径和相关功能基因的鉴定奠定了基础。
目前,萜类物质生物合成途径中关键酶基因的研究主要集中在前体合成阶段,即通过MEP途径和MVA途径合成IPP和其异构体DMAPP,后半段(骨架构建和后修饰)生物合成途径的研究,在萜类方面尤其是环烯醚萜类的研究还很少,青叶胆的基因克隆与功能基因鉴定还处于空白状态。基于此,本研究通过实时荧光定量PCR测定了不同发育时期候选关键酶基因的表达量。
材料:按苗期、花期、果期于云南省红河州弥勒市采集青叶胆全株。经云南中医学院刘小莉副教授鉴定。
试剂:柱式植物总RNA抽提纯化试剂盒(批号:SK8661)、SG Fast qPCR Master Mix(High Rox,批号:B639273)均购自生工生物工程(上海)有限公司;Thermo ScientificTMRevertAid Premium Reverse Transcriptase试剂盒(ABI,批号:EPO773)。
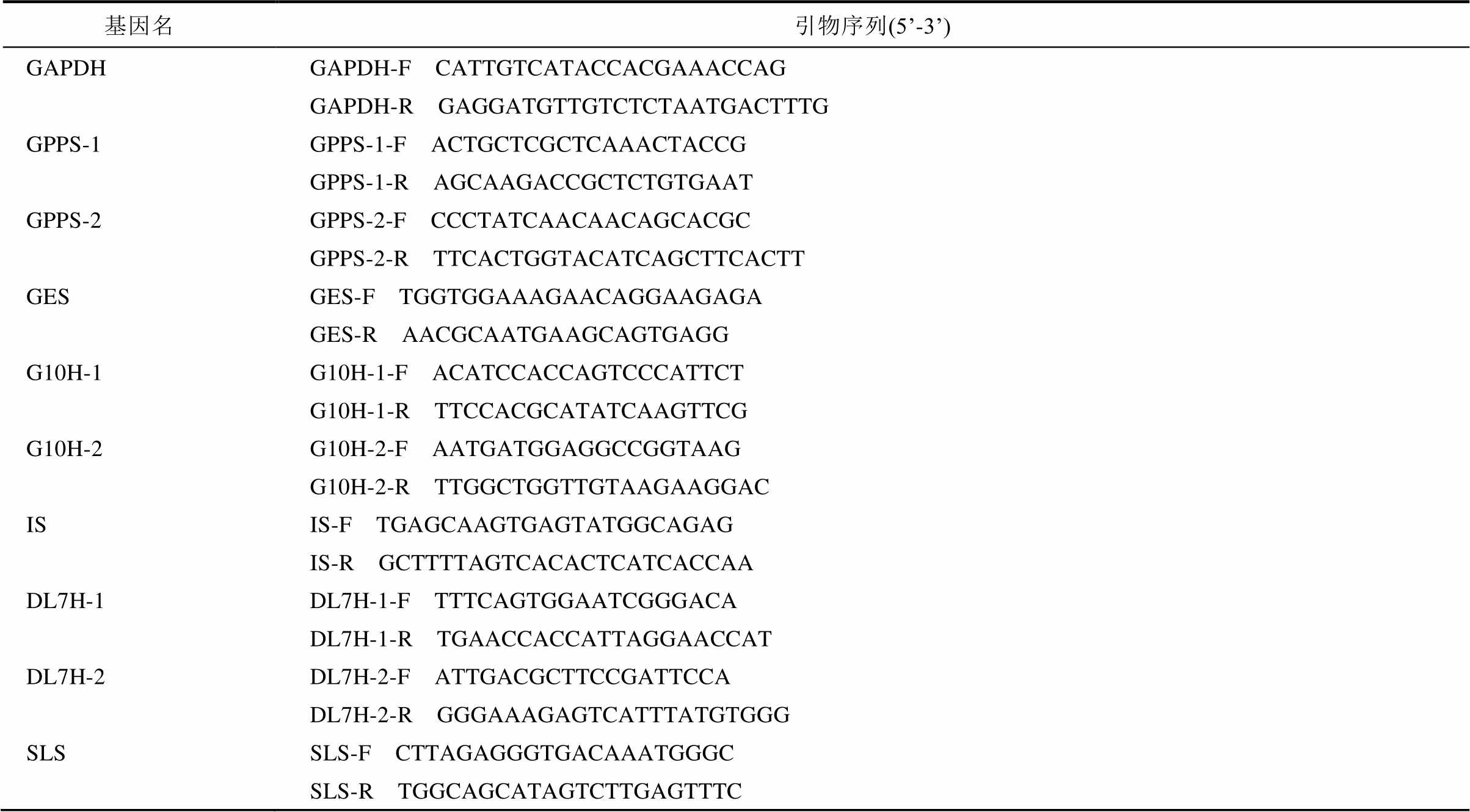
根据青叶胆转录组数据中挖掘得到的关键酶基因序列,利用Primer Premier 5.0设计实时荧光定量PCR引物,并以GAPDH为内参基因,引物序列见表4。
表4 荧光定量PCR引物
Tab. 4 Designed Primer of quantitative Real-time PCR
利用柱式植物总RNA抽提纯化试剂盒提取苗期、花期、果期青叶胆全株的RNA,利用Thermo Scientific™ RevertAid Premium Reverse Transcriptase试剂盒将RNA反转录为cDNA。取已制备好的10个基因对照品,稀释10倍,用于制作标准曲线。反应体系为20 µL,包含正反引物各0.4 µL,SybrGreen qPCR Master Mix 10 µL,ddH2O 7.2 µL,cDNA模板2 µL。每个样品做3次重复和1个没有模板的阴性对照,将96孔板按上述加好样品后放入荧光定量PCR仪,反应条件为:95 ℃ 3 min,1个循环;95 ℃ 7 s,57 ℃ 10 s,72 ℃ 15 s,45个循环。
将10个基因的标准曲线和与检测各样品后得到的Ct值相结合,计算每个样品的定量结果,数据分析采用法2-DDCt法。
GPPS-1、GPPS-2、GES、G10H-1、G10H-2、IS、DL7H-1、DL7H-2和SLS在青叶胆不同发育时期的变化如图4所示,各基因在青叶胆不同发育时期均有表达,且除SLS以外,其余8个关键酶基因的表达规律相似,最高表达量均出现在青叶胆的果期,花期次之,最低表达量均出现在青叶胆的苗期。所不同的是SLS的表达规律,最高表达量出现在花期,果期次之,苗期最低。通过实时荧光定量PCR,测定了不同发育时期獐牙菜苦苷生物合成途径骨架构建和后修饰9个候选关键酶基因的表达规律,为后续深入开展基因克隆、功能验证等奠定了基础。
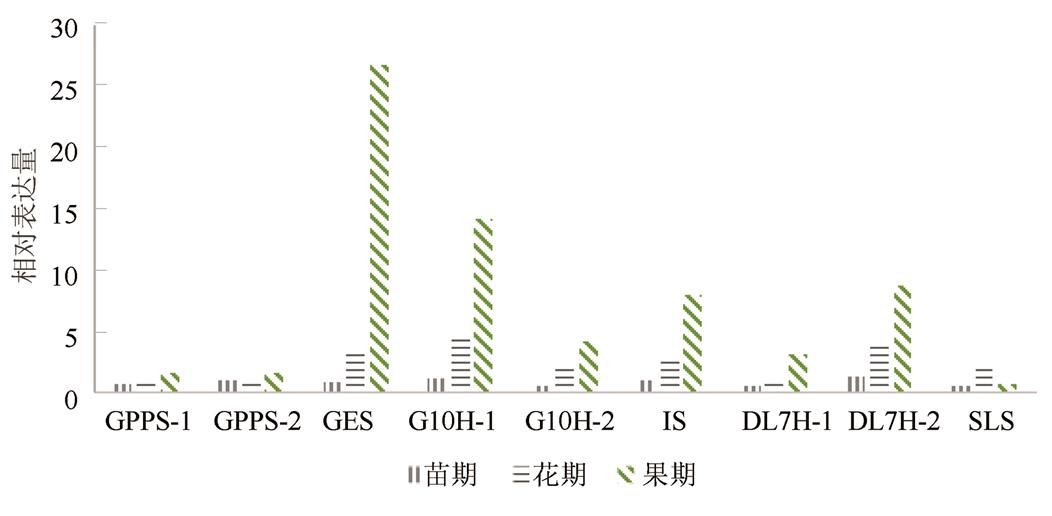
图4 不同发育时期青叶胆酶基因的表达分析
Fig. 4 Expression and analysis of relative enzymes in different development period ofS. mileensis
本研究首次采用高通量转录组测序技术对青叶胆全草进行了高通量测序,拼接获得了68 643条青叶胆Unigene,N50平均长度1 325 bp,共有42 954条Unigene得到注释。与其他已测序的物种相比,如草麻黄的N50平均长度为553 bp[19],文冠果的N50的平均长度为1 180 bp[20],青叶胆转录组的组装结果好,完整性较高。同时找到了参与萜类前体生物合成的MVA和MEP途径的Unigene 43条,催化IPP和DMAPP缩合成香叶基焦磷酸(GPP)的Unigene 3条,发现了可能参与獐牙菜苦苷生物合成最后修饰阶段的Unigene 25条,并且对獐牙菜苦苷生物合成后半段9个候选关键酶基因进行了表达规律的研究,为后续深入开展青叶胆的研究提供数据支撑,为青叶胆功能基因的发掘利用、特异miRNA的功能分析及基因组测序等奠定基础。
REFERENCES
[1] 中国科学院中国植物志编辑委员会. 中国植物志[M]. 第62卷. 北京: 科学出版社, 1988: 392.
[2] 中国药典. 一部[S]. 2015: 196.
[3] 陈纪军, 耿长安. 云南特有抗肝炎中药青叶胆中系列新奇骨架内酯成分与抗乙肝病毒活性[C]. 全国天然药物资源学术研讨会. 2010.
[4] WANG M R, HUANG H Y. Study on germination from the different mating patterns inSwertia mileensis[J]. Chin J Ethnomed Ethnopharm(中国民族民间医药), 2015(2): 10-11.
[5] 肖琳. 青叶胆化学成分研究及2010版中国药典青叶胆标准修订研究[D]. 陕西: 西北大学, 2009.
[6] CHEN K, WU T, SONG H P. Research progress on pharmacological activities of swertiamarin [J]. Drugs Clin(现代药物与临床), 2016, 31(10): 1684-1687.
[7] LI S X, CHEN L Y, XIA C L. Analysis of genetic diversity in certifiedSwertia mileensisby ISSR [J]. Lishizhen Med Mater Med Res(时珍国医国药), 2015, 26(7): 1748-1749.
[8] HUANG H Y, HUANG J, WANG M R, et al. Comparison on progeny regeneration capacity among different mating patterns and optimization of cultivation condition inSwertia mileensis[J]. Chin Tradit Herb Drugs(中草药), 2016, 47(3): 480-487.
[9] YANG Y H, YANG L F, FAN J, et al. Strategy on persistent utilization ofSwertia mileensis[J]. Chin J Ethn Ethn(中国民族民间医药), 2003(61): 107-109.
[10] 耿长安. 抗肝炎中药青叶胆的抗乙肝病毒活性成分研究 [D]. 中国科学院研究生院, 2011.
[11] OKSMANCALDENTEY K M, INZÉ D. Plant cell factories in the post-genomic era: new ways to produce designer secondary metabolites [J]. Trends Plant Sci, 2004, 9(9): 433-440.
[12] XIAO M, ZHANG Y, CHEN X, et al. Transcriptome analysis based on next-generation sequencing of non-model plants producing specialized metabolites of biotechnological interest [J]. J Biotechnol, 2013, 166(3): 122-134.
[13] ZOU L Q, KUANG X J, LI Y, et al. Advance in transcriptomic studies of ginseng species [J]. Chin J Chin Mat Med(中国中药杂志), 2016, 41(22): 4138-4143.
[14] GUO X, LI Y, LI C, et al. Analysis of theDendrobium officinaletranscriptome reveals putative alkaloid biosynthetic genes and genetic markers [J]. Gene, 2013, 527(1): 131-138.
[15] LI Y, SUN C, LUO H M, et al. Transcriptome characterization forSalvia miltiorrhizausing 454 GS FLX [J]. Acta Pharm Sin(药学学报), 2010, 45(4): 524-529.
[16] 吉雪琪. 地黄中梓醇含量变化及萜类合成关键酶基因表达的初步研究[D]. 北京: 北京协和医院, 2014.
[17] 王俊峰. 川西獐牙菜体细胞杂交及其环烯醚菇类化合物合成相关基因香叶醇-10-羟化酶的克隆和功能验证[D]. 山东: 山东大学, 2010.
[18] 王彩云, 张晓东, 沈涛, 等. 龙胆苦苷生物合成途径研究进展[J]. 江苏农业科学, 2014, 42(3): 4-10.
[19] MA J, CHENG T L, SUN C Y, et al. Characterization of transcriptome reveals pathway of flavonoids inEphedra sinicaStapf [J]. Acta Agricul Zhejiangensis(浙江农业学报), 2016, 28(4): 609-617.
[20] AO Y, MA L Y, HAN S W, et al. Transcriptome analysis forXanthoceras sorbifoliabunge based on high-throughput sequencing technology [J]. J China Biotechnol(中国生物工程杂志), 2015, 35(7): 22-29.
(本文责编:李艳芳)
Analysis of the Transcriptome ofSwertia Mileensisbased on High-throughput Sequencing
WU Xinyi, YAN Yuan, LIU Xiaoli*
(College of Traditional Chinese Medicine, Yunnan University of Traditional Chinese Medicine, Kunming 650500, China)
ABSTRACT: OBJECTIVETo explore the genetic basis of the biosynthesis of the swertiamarinin inS. mileensis.METHODSThe high throughput sequencing by Illumina Hiseq 2500 was used to sequence its transcriptome ofS. mileensis.RESULTSA total of 685 643 unigenes were assembled with an average length of 901 bp. Among them, 42 954 unigenes were annotated in the public databases NR, SwissProt, COG, KOG, GO and Pfam. The results showed that 43 unigenes were involved in the production of intermediate, 3 unigenes catalyze synthesis of terpenoids skeleton, and 25 unigenes might participate in terpene post-modification in the three steps of biosynthesis of swertiamain.CONCLUSIONThis study is the first comprehensive study on transcriptome ofS. mileensis, and candidate genes involves in the biosynthesis of swertiamarin are obtained. The transcriptome data constitutes a much more abudant genetic resource that can be utilized to benefit further molecular biology studies onS. mileensis.
KEY WORDS:Swertia mileensis; transcriptome; Illumina Hiseq 2500; swertiamain
中图分类号:R915
文献标志码:B
文章编号:1007-7693(2018)03-0363-07
DOI:10.13748/j.cnki.issn1007-7693.2018.03.012
引用本文:吴昕怡, 严媛, 刘小莉. 基于高通量测序的青叶胆转录组研究[J]. 中国现代应用药学, 2018, 35(3): 363-369.
收稿日期:2017-08-29
基金项目:国家自然科学基金(81660634);云南省科技计划项目(2014FZ077)
作者简介:吴昕怡,女,硕士生 Tel: (0871)65918129 E-mail: 2665438954@qq.com
*通信作者:刘小莉,女,博士,副教授 Tel: (0871)65918129 E-mail: kmxunzi@aliyun.com